Uso de modelos simplificados para la predicción teórica de las interacciones entre los anticuerpos disponibles y el dominio de unión al receptor de la proteína espiga del SARS-CoV-2
G. A. Huerta-Miranda , W. I. García-García , A. Vidal-Limon and M. Miranda-Hernández - JOURNAL OF BIOMOLECULAR STRUCTURE AND DYNAMICS
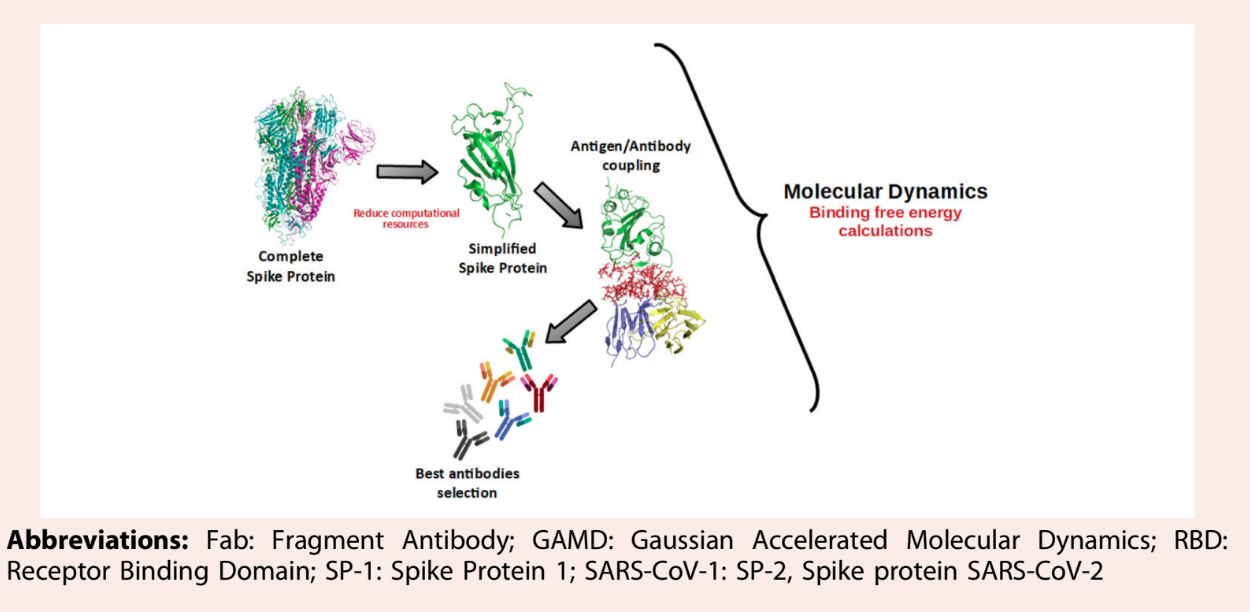
El impacto negativo de enfermedades infecciosas como el COVID-19 en la salud pública y la economía global es evidente. Esta pandemia representa un desafío importante para que la comunidad científica desarrolle nuevos métodos analíticos prácticos para diagnosticar con precisión los casos emergentes. Debido a su selectividad y sensibilidad, se hacen necesarias nuevas metodologías basadas en interacciones antígeno/anticuerpo para detectar biomarcadores de COVID-19. En este contexto, el modelado computacional teórico reduce los esfuerzos experimentales y ahorra recursos para el diseño racional de biosensores. Este estudio propone el uso de la dinámica molecular para predecir las interacciones entre el dominio de unión al receptor (RBD) del modelo simplificado de la proteína del pico del SARS-CoV-2 y un conjunto de anticuerpos altamente caracterizados. La energía libre de unión de los complejos antígeno/anticuerpo se calculó para los modelos simplificados y se comparó con el ectodominio completo del SARS-CoV-2 para validar la metodología. Los datos estructurales derivados de nuestros cálculos de dinámica molecular y energía libre de punto final mostraron una correlación positiva entre ambas aproximaciones, con un coeficiente de correlación de Pearson de 0,82;t = 3,661, df = 3, valor de p = 0,03522, con un intervalo de confianza del 95 %. Además, identificamos los residuos interfaciales que podrían generar enlaces covalentes con una superficie química específica sin perturbar la dinámica de enlace para desarrollar dispositivos de diagnóstico altamente sensibles y específicos. Comunicado por Ramaswamy H. Sarma.nnPalabras clave: COVID-19; SARS-CoV-2; Modelos simplificados; energía libre vinculante; dinámica molecular.
